023-67630383
 销售咨询QQ2548969917
销售咨询QQ2548969917 技术咨询QQ1291769782
技术咨询QQ1291769782热线/微信:13681365274
 销售咨询QQ2548969917
销售咨询QQ2548969917 技术咨询QQ1291769782
技术咨询QQ1291769782微信扫一扫
下载说明书


Massively parallel Cas13 screens reveal principles for guide RNA design - PMC
VI型CRISPR酶最近被鉴定为可编程RNA指导的RNA靶向Cas蛋白,具有核酸酶活性,允许在不改变基因组的情况下敲除靶基因。除了靶RNA敲除,Cas13蛋白还被用于病毒RNA检测,定点RNA编辑,m6A修饰转录物的去甲基化,RNA活体成像,剪接位点选择的调节以及切割和聚腺苷酸化位点的使用。Cas13蛋白通过单个CRISPR RNA (crRNA)被引导至其靶RNA,CRISPR RNA由直接重复(DR)茎环和间隔序列(引导RNA或gRNA)组成,间隔序列通过RNA-RNA杂交介导靶识别。虽然Cas13酶在激活时发挥一些非特异性的并行核酸酶活性,但与RNA干扰相比,它们在培养细胞中大大降低了脱靶活性。先前的研究表明,Cas13 gRNAs具有最小的原间隔区侧翼序列(PFS)限制,并且Cas13结合的RNA靶位点应该是可接近的。除了这些基本参数,我们目前缺乏关于有效靶RNA敲除的最佳Cas13 crRNA设计的信息。
迄今为止,已有三种Cas13效应蛋白(PguCas13b、PspCas13b、RfxCas13d)被报道显示出高RNA敲除效率,且脱靶活性最小。我们比较了这些Cas13酶在指向胞质溶胶或细胞核时敲低GFP mRNA的能力。RfxCas13d (CasRx)始终显示最强的靶基因敲除,特别是当融合到核定位序列(NLS)时(补充图1a–c)。使用Cas13d-NLS,我们改变了gRNA长度,同时相对于30 nt参照gRNA保持恒定的gRNA 5’端或3’端,并发现23–30nt grna赋予最显著的靶敲减(补充图1d)。
为了系统地评估数千种gRNAs的RfxCas13d靶向敲除功效,我们建立了一个表达不稳定GFP和多西环素诱导的Cas13d-NLS核酸酶的单克隆HEK293细胞系。我们慢病毒递送了7500个靶向GFP编码序列的crRNAs文库,包含完全匹配和错配gRNAs(图1a)。我们基于它们的GFP强度对四个箱中的门控细胞进行荧光激活细胞分选(FACS )(补充图2a)。指导RNA计数包含具有最低GFP表达的细胞(补充图2 b–d)。
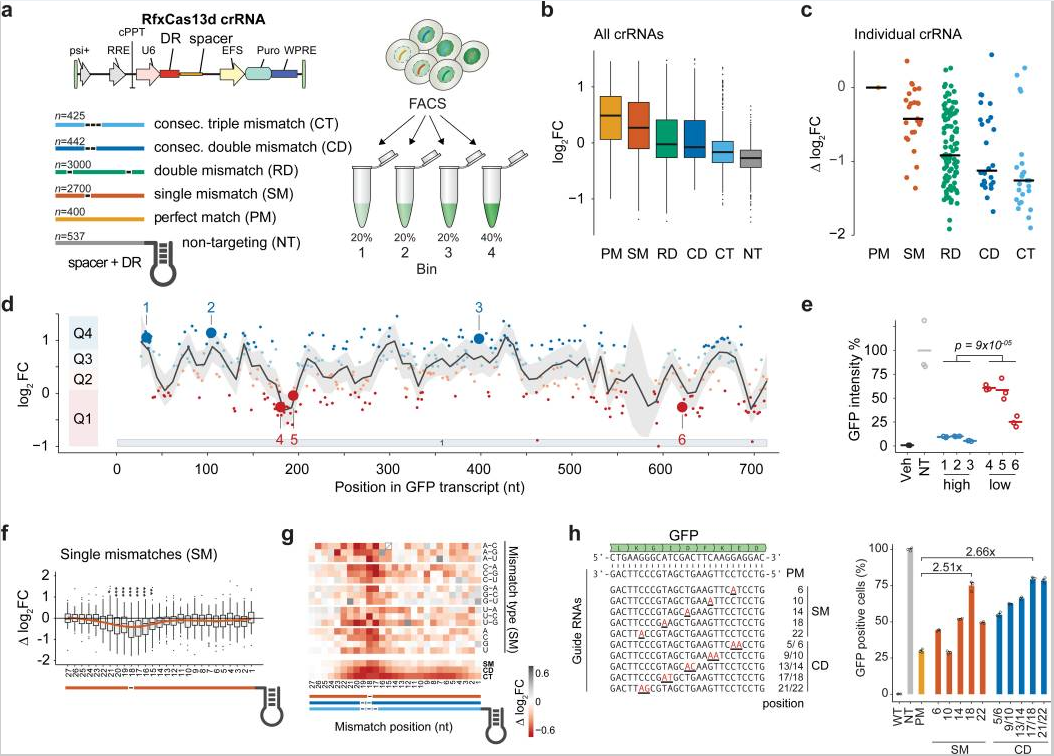
我们计算了所有箱之间的log2倍变化(log2FC) gRNA富集和未分类的输入gRNA分布(补充数据1)。完全匹配的gRNA在bin 1中富集,而错配数量的增加导致gRNA富集的逐渐减少(图1b,补充图3a-c)。对于整个gRNA群体以及单个grna和它们相应的具有1-3个错配的grna都是如此(图1 b-c,补充图3d)。作为对照,该文库还包含537个非靶向crRNAs,它们被有效地从bin 1中去除(图1b,补充图3a-c)。正如所预期的,仓1中的gRNA丰度与仓2至4中的负相关,仓2至4包含具有更高GFP强度的细胞(补充图3e,f)。总之,这表明bin 1中grna的富集准确地反映了靶mRNA的敲除。
我们注意到在每个gRNA类中gRNA富集的相当大的异质性(图1 b–c)。对于靶向靶mRNA不同区域的完全匹配gRNA,我们观察到位置依赖性效应,表明靶序列背景对gRNA功效的影响(图1d)。我们沿着GFP靶转录物选择了6个高或低富集的grna,并通过转染单个grna随后进行流式细胞术来验证它们的相对靶敲除效率(图1e)。
为了检验Cas13是否能耐受gRNA和靶RNA之间的错配,我们通过从参照(完全匹配)gRNA中减去log2FC来计算每个错配gRNA的相对log2倍变化(δlog2FC)(图1f)。我们在gRNA核苷酸15至21之间发现了Cas13d敲除效力的关键(“种子”)区域,其中心位于相对于gRNA 5’端的核苷酸18。尽管已经显示了Cas13a的种子区域,但一组报道没有Cas13d的清晰种子区域23,而另一组在无细胞测定中显示了Cas13d的位置依赖性错配敏感性。在种子区域内,单错配导致gRNA富集减少,而种子区域外的错配被更好地耐受(图1f)。临界区的存在与错配身份无关(图1g)。类似地,连续的双重和三重错配表明存在临界区(图1g,补充图4a)。对于随机分布的双错配,在两个错配都在种子区域的情况下观察到富集的最大变化(补充图4b)。将错配的数量增加到三个大大取消的靶敲除(补充图4a)。由于这个原因,关键区域可能在之前测试四个连续错配的EsCas13d研究中被掩盖了。
考虑到在种子区域具有错配的gRNAs的富集的异质性,我们试图评估周围核苷酸环境的影响(补充图5a)。对照参考gRNA功效,靶位点的“U”背景中的错配对Cas13d活性有负面影响,而“GC”背景中的错配有更好的耐受性(补充图5b)。我们在使用与GFP mRNA具有单或双核苷酸错配的grna的转染实验中证实了种子区域的存在(图1h)。虽然完全匹配的gRNA将GFP阳性细胞的百分比降低至约29%,但在gRNA位置18的单一错配导致75%的GFP阳性细胞,在位置17和18的双重错配导致约79%的GFP阳性细胞(图1h)。
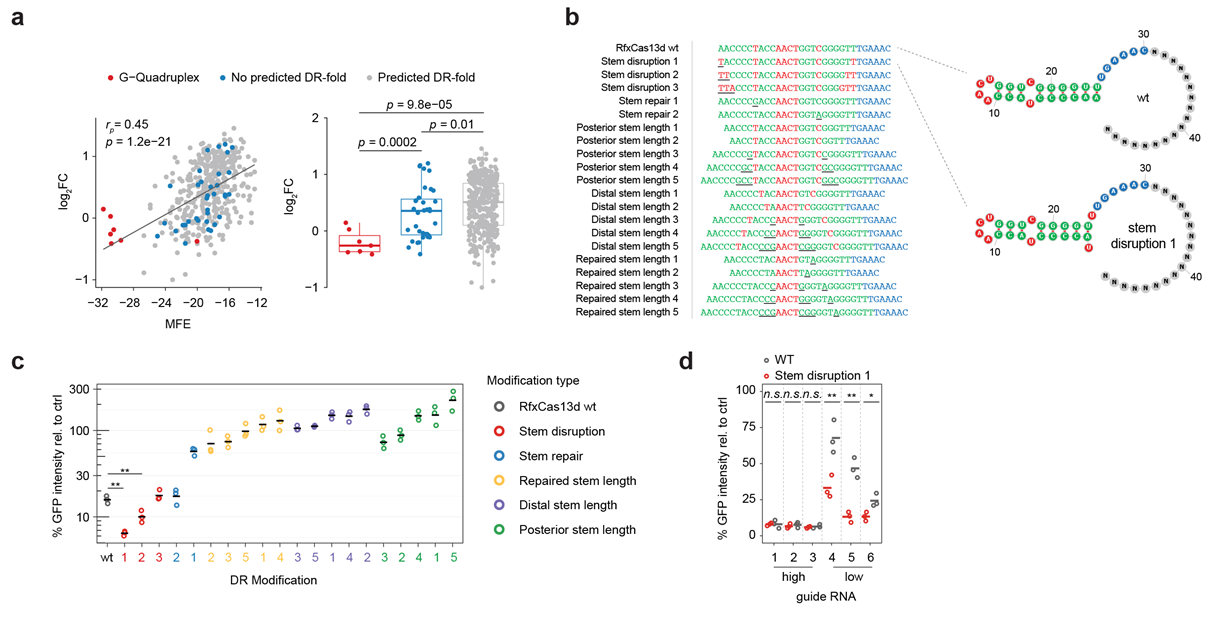
接下来,我们试图评估可能影响完全匹配gRNAs的敲除功效的特征(详见补充注释1)。影响在GFP平铺屏幕中观察到的gRNA富集的特征之一是crRNA折叠:完全匹配crRNA的预测二级结构和相应的最小自由能(MFE)显示了MFE和gRNA功效之间的正相关性(补充图6a)。特别是,依赖G的结构,如预测的G-四链体,显示出靶基因敲除的减少。考虑到crRNA折叠对于有效的靶基因敲除是至关重要的,我们试图通过修复DR中预测的凸起、改变茎环的长度或破坏近端DR茎中的碱基来进一步稳定和改善DR(补充图6b)。对EsCas13d和UrCas13d的晶体结构及其crRNA的分析表明,DR中的末端环可能没有嵌入蛋白质中,因此可能允许茎环23,24的延伸(和进一步的稳定),类似于先前发现的增强Cas9活性的那些25,26。我们观察到茎长度的任何变化都完全消除了靶敲除(补充图6c)。此外,修复茎内凸出的核苷酸减少了靶基因的敲除。然而,破坏近端茎内的第一个碱基对进一步增加了Cas13d的靶向效力,导致了一种新的RfxCas13d DR,具有改善的敲除。我们在另外6种靶向GFP的grna上测试了修饰的DR,发现修饰的DR改善了靶基因敲除,特别是对于具有低敲除功效的grna(补充图6d)。
我们根据与观察到的gRNA富集的相关性定义了15种crRNA和靶RNA特征(补充表1,补充注释1)。利用这些特征,我们试图推导出一个可概括的“靶上”模型来预测Cas13d靶敲除。我们比较了机器学习方法预测gRNA功效的能力(见方法),并发现随机森林(RF)模型具有最佳的预测准确性(补充图7a),将crRNA折叠能量、局部靶‘C’上下文和上游靶‘U’上下文作为最重要的特征进行加权(补充图7b)。其他学习方法经常选择类似的特征,表明这些特征是Cas13d GFP敲低的主要驱动因素(补充图7c)。为了确定gRNA疗效的关键预测因子,我们反复减少特征的数量,监测模型性能,并推导出一个最小模型,该模型解释了约37%的方差(r2 ),与保留数据的Spearman相关(rs)约为0.58(图2a,补充图7d–f)。相比之下,具有与Cas9 gRNA预测算法27类似结构的支持向量机(SVM)回归模型在应用于该数据时表现更差(r2 = 0.21,rs = 0.44)(图2a)。
为了表明我们的模型是可推广的,我们预测gRNAs靶向编码细胞表面蛋白的CD46和CD71的内源性转录物,并通过流式细胞术测量gRNA敲除的功效。对于每个基因,我们选择了3个预测具有高敲低功效的grna(前2个四分位数)和3个预测具有低敲低功效的grna(后2个四分位数)。在个体gRNA水平上,我们发现大多数具有较高预测指导分数的grna比具有较低指导分数的grna更强有力地抑制CD46和CD71蛋白表达(图2b)。比较观察到的所有三个高评分gRNAs与所有三个低评分gRNAs的敲除,我们发现CD71有显著改善,而CD46则观察到相当大的差异。为了增加通量并测试更多基因的gRNA功效预测,我们首先产生了一个小的crRNA文库,该文库以10个必需基因和10个对照基因为目标,具有3个高分和3个低分grna,并随着时间的推移在基因必需性筛选中监测它们的损耗。从先前RNAi筛选28中强烈缺失的基因中选择必需基因(补充图8a)。大多数靶向必需基因的高评分grna随着时间的推移逐渐耗尽,而低评分grna基本上没有耗尽(图2c,补充图8b)。
为了表明我们的模型是可推广的,我们预测gRNAs靶向编码细胞表面蛋白的CD46和CD71的内源性转录物,并通过流式细胞术测量gRNA敲除的功效。对于每个基因,我们选择了3个预测具有高敲低功效的grna(前2个四分位数)和3个预测具有低敲低功效的grna(后2个四分位数)。在个体gRNA水平上,我们发现大多数具有较高预测指导分数的grna比具有较低指导分数的grna更强有力地抑制CD46和CD71蛋白表达(图2b)。比较观察到的所有三个高评分gRNAs与所有三个低评分gRNAs的敲除,我们发现CD71有显著改善,而CD46则观察到相当大的差异。为了增加通量并测试更多基因的gRNA功效预测,我们首先产生了一个小的crRNA文库,该文库以10个必需基因和10个对照基因为目标,具有3个高分和3个低分grna,并随着时间的推移在基因必需性筛选中监测它们的损耗。从先前RNAi筛选28中强烈缺失的基因中选择必需基因(补充图8a)。大多数靶向必需基因的高评分grna随着时间的推移逐渐耗尽,而低评分grna基本上没有耗尽(图2c,补充图8b)。
此外,我们在A375细胞中进行了第二次靶向必需性筛选,靶向35个必需基因和65个对照基因,其中有20个高评分和20个低评分gRNAs。类似于上面的HEK293筛选,我们发现靶向必需基因的高分gRNAs随着时间推移而逐渐耗尽(图2d,补充图8c)。虽然在每个基因水平上,高评分grna通常比低评分grna更耗尽,但我们注意到,并非所有预测的必需基因在Cas13d靶向时都表现出耗尽(补充图8c-d),这表明RNAi筛选得出的必需性得分可能无法直接与Cas13得出的必需性得分相比较。
我们基于20个高评分gRNA的gRNA等级一致性计算了基因缺失的显著性得分,并在列表顶部发现了定义的必需基因的强烈富集(图2e)。与Cas9 STARS得分29(高达rs = 0.61)相比,gRNA消耗得分与用于定义待测必需基因组的DEMETER2 RNAi 28得分的相关性更好(使用最佳gRNA高达rs = 0.71)(图2f)。综上所述,这表明来源于GFP平铺筛选的crRNA和靶RNA特征可以概括预测新靶的Cas13d gRNA功效,并且这些gRNA预测可以用于合并的CRISPR-Cas13筛选。
我们基于GFP平铺屏幕的预测性靶标模型很大程度上能够将低敲除功效的gRNAs与高功效的gRNAs分开。然而,鉴于我们在预测的高分gRNAs中观察到剩余的异质性,我们试图通过扩展我们的训练数据集来改进我们的目标模型。因此,我们对HEK293细胞中细胞表面蛋白CD46、CD55和CD71的主要转录亚型进行了三次额外的Cas13d平铺筛选,结合FACS选择表面蛋白表达降低的细胞(图3a–c;补充图9a–c)。除了完美匹配gRNA,我们还添加了几个额外的gRNA类(补充图9a)。对于每个筛选,相对于未分类的输入样品,完全匹配的gRNA显示最强的gRNA富集,而反向补体阴性对照grna和非靶向grna被耗尽(补充图9d)。在新的筛选中,我们将总gRNA长度减少到23个碱基,并包括一组长度为15-36个核苷酸的gRNA长度变体。从23个核苷酸长度开始,gRNAs发挥完全的敲除功效,而更长的gRNA 3’末端没有任何有害作用。
与靶向非翻译区(UTRs)或内含子的grna相比,靶向编码区(CDS)的完全匹配grna更加富集(补充图9f)。UTR靶向gRNAs可能表现出较低的富集度,因为每个靶基因可能由多个转录物同种型代表,并交替使用UTR。因此,靶向编码区的grna找到同源靶位点的可能性更高,而例如,靶向3’UTR的grna仅在一小部分表达的转录物同种型中找到它们的靶位点。因此,内含子靶向gRNAs的低富集可以用内含子的短命性质来解释。对于这些gRNAs,内含子靶位点只存在很短一段时间,这可能使转录物逃避Cas13靶向。由于这个原因,靶向CDS的gRNA和靶向UTR或内含子的grna之间的gRNA敲除效率可能不是直接可比的。在所有39个内含子中,我们发现以内含子为目标的gRNAs仅轻度富集。在这些内含子中,我们观察到紧接5’-剪接位点下游和3’-剪接位点上游50到0个核苷酸内的gRNA功效轻微降低(补充图9g)。这些位点通常被剪接体30结合,表明靶向这些区域的gRNAs可能与剪接机制和其他剪接因子竞争靶序列。由于核内转录物的成熟似乎影响gRNA的靶向效力,我们想知道外显子-连接-复合物(EJC)是否会以同样的方式影响成熟转录物的敲除。在剪接31,32时,EJC通常结合外显子-外显子-连接处上游约20-24个核苷酸。事实上,我们在外显子连接处5’端的20到0个核苷酸的窗口内观察到高评分gRNAs的缺失(补充图9h)。
为了改进我们的靶向模型,我们专注于靶向CDS区域的完美匹配gRNAs,并将高置信度模型输入观察值的数量从大约400增加到近3000。类似于最初的GFP筛选,gRNAs功效以非随机方式沿编码区分布(图3a–c)。我们重复了对可能影响敲除功效的特征的评估(详见补充注释2)。值得注意的是,观察次数的增加揭示了位置核苷酸偏好(补充图10a–b)。在gRNA第18位附近的种子区域,指导RNA的富集与G-和C-碱基的概率正相关。在这个区域周围,U-和A-碱基概率与目标敲低正相关。我们使用所有四个平铺屏幕上的2918个CDS靶向gRNAs推导出一个更新的靶向模型,并以与之前类似的方式从644个评估特征中选择35个(见方法)(补充表2、补充注释2、补充数据2)。
与初始RFminimal模型(此后称为RFGFP)相比,组合随机森林模型(RFcombined)显示出改进的预测准确性,解释了约47%的方差(r2 ),与保留数据的Spearman相关性(rs)为约0.67(图3d,补充图10c)。使用10倍交叉验证,该模型有效地将低评分grna与高评分grna分开,将63%的grna正确地分配到最高功效四分位数(图3e)。类似地,排名靠前或靠后的grna的预测指导分数(通过观察到的敲除功效排名)将表现良好的grna与表现差于预期的grna分开(补充图10d)。此外,我们对三个数据集进行留一交叉验证训练,同时预测保留的第四个屏幕的指南分数。RF组合模型很好地概括了内源基因(平均SD:RS = 0.63±0.01),但对GFP转基因的预测性较低(rs = 0.33)(补充图10e)。
最后,我们比较了RFGFP和RFcombined两种模型正确预测两种必需性筛选的击倒效果的能力。两个屏幕都是基于RFGFP模型所做的gRNA预测而设计的。在这两种情况下,RFcombined与观察到的所有基因的敲除功效更一致(图3f)。同样,我们发现,对于A375适应性筛选中10个最缺失的基因,RFcombined显示出与观察到的gRNA缺失在基因水平上的一致性提高(RF GFP:RS = 0.46±0.16,RF combined:0.58±0.14)。总之,我们表明我们更新的RFcombined on-target模型能够预测Cas13d gRNA靶敲除功效,将表现不佳的gRNA与功效较高的grna分开,并推广到众多靶点。
我们应用我们的模型预测了人类基因组中所有蛋白质编码转录物的gRNAs。我们通过一个用户友好的基于网络的应用程序(https://cas 13 design . ny genome . org)提供了这些预测。此外,我们报告了每个转录物的5’UTR、CDS和3’UTR的10个最高得分的crRNAs(补充图11a,补充数据3)。我们根据四个筛选中的功效四分位数对预测的gRNAs进行了划分。在所有可能的gRNAs中,只有15.2%属于得分最高(最佳敲除)的四分位数(Q4)(补充图11b)。大部分gRNA被预测具有较低的功效(所有grna的36.8%在Q1或Q2),这强调了高敲除功效的最佳gRNA选择的价值。然而,几乎所有的转录物都含有得分最高的gRNAs(补充图11c)。
总之,我们为CRISPR-Cas13d执行了一组合并筛选,并为最佳gRNA设计定义了目标规则。我们证明了crRNA的特征和靶RNA的环境限制了靶基因敲除的效率,并且利用这些数据,我们开发了一个模型来预测具有高效能的grna。我们使用合并的Cas13d筛选验证了该模型,并比较了Cas13扰动与先前RNAi和Cas9筛选识别一组必需基因的能力。虽然所有三种扰动方法大体一致,但重要的是要注意到一个全面的全基因组比较尚待进行。RNA靶向方法之间的一个重要区别是,虽然RNAi局限于胞质溶胶,但Cas13允许区域化靶向(细胞核、胞质溶胶和其他亚细胞区室)和复杂的转录组工程与催化死亡(dCas13)效应子融合。总之,我们的研究提供了Cas13靶向的详细特征和高活性grna的预测模型,为大规模并行RNA靶向筛选的设计提供了一个有价值的平台。