023-67630383
 销售咨询QQ2548969917
销售咨询QQ2548969917 技术咨询QQ1291769782
技术咨询QQ1291769782热线/微信:13681365274
 销售咨询QQ2548969917
销售咨询QQ2548969917 技术咨询QQ1291769782
技术咨询QQ1291769782微信扫一扫
下载说明书


iStop CRISPR基因敲除技术(1)与以往的CRISPR Cas9基因敲除技术的不同之处在于,不需要DNA双链断裂即可实现基因敲除。iStop技术可以在基因组上实现C>T突变,精确地将四个密码子(CAA、CAG、CGA 和 TGG)转换为终止密码子。由于不需要双链DNA断裂,基因编辑精确温和,对细胞影响极小,最大限度保存了细胞的状态和功能。
我们采用该技术对稳转了mcherry细胞的293T细胞株中的mcherry报告基因进行了敲除。结果显示,iStop技术基因敲除效率高,编辑精确可控,可以方便地用于细胞基因敲除。
iStop基因敲除原理
无双链DNA切割活性的Cas9突变体连接了APOBEC酶,可以将靶点的特定位置核苷酸中的C突变为T,从而将将四个密码子(CAA、CAG、CGA 和 TGG)转换为终止密码子(图1)。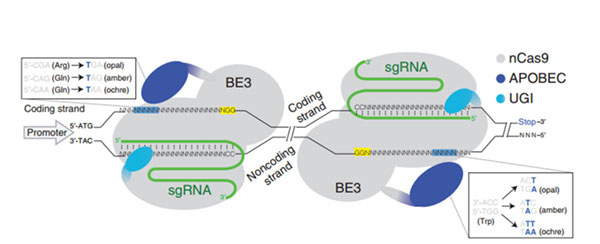
图1 iStop技术原理(图片来源:Kuscu, Cem, et al. "CRISPR-STOP: gene silencing through base-editing-induced nonsense mutations." Nature methods 14.7 (2017): 710-712.)
1、设计mcherry基因敲除靶点
为了直观地检测基因敲除效果,我们选择了293V-mcherry细胞株。该细胞稳定整合mcherry红色荧光蛋白,并且是单克隆细胞株,细胞荧光亮度均一,能比较直观的观测到基因敲除效。顺便推荐我们的293V-mcherry细胞株(货号CG013),真的荧光明亮,亮度均一。
根据mcherry荧光蛋白的编码序列,我们设计了3个基因敲除靶点(图2)。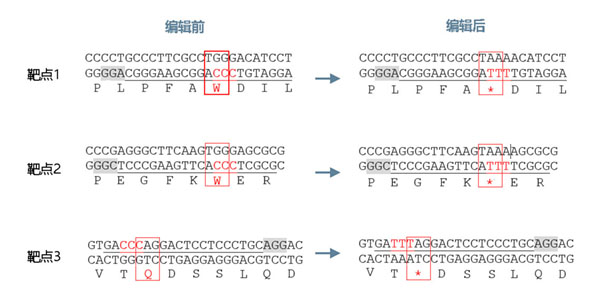
图2 mcherry基因敲除靶点设计
2、体外酶切验证靶点效率
虽然iStop基因敲除不需要双链DNA断裂,但是sgRNA与Cas9蛋白的结合以及形成Cas9蛋白:sgRNA:DNA复合物与Cas9切割DNA的前期是一样的。我们推测,采用Cas9体外酶切试剂盒对靶点进行验证,也可以一定程度上预测该靶点在细胞内iStop基因编辑的效率。
我们采用sgRNA合成试剂盒(英茂盛业,PC1380)合成了3个mcherry靶点。PCR扩增mcherry及附近序列。采用spCas9体外酶切试剂盒(英茂盛业,PC1400)对靶点进行了酶切验证。结果显示,靶点1、3切割效果较好,靶点2较差(图3)。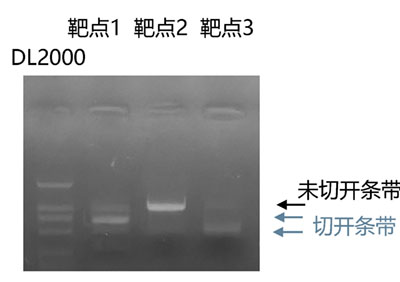
图3 体外酶切验证靶点
3、构建dCas9-BE3表达载体和3个靶点sgRNA表达载体
为了方便显示转染成功的细胞,我们在Cas9nick-BE3表达载体上加入了GFP荧光标签。
载体图谱如图4所示:
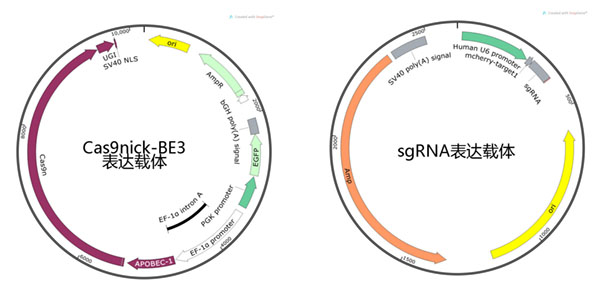
图4 载体图谱
4、转染及检测
293-mcherry细胞接种到24孔细胞培养板,24小时后将上述两个载体按照下表转染。转染后72小时取部分细胞进行基因组PCR检测,剩余细胞重新贴壁后进行荧光显微镜观察。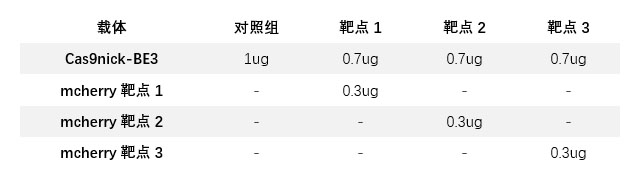
4.1、荧光显微镜检测结果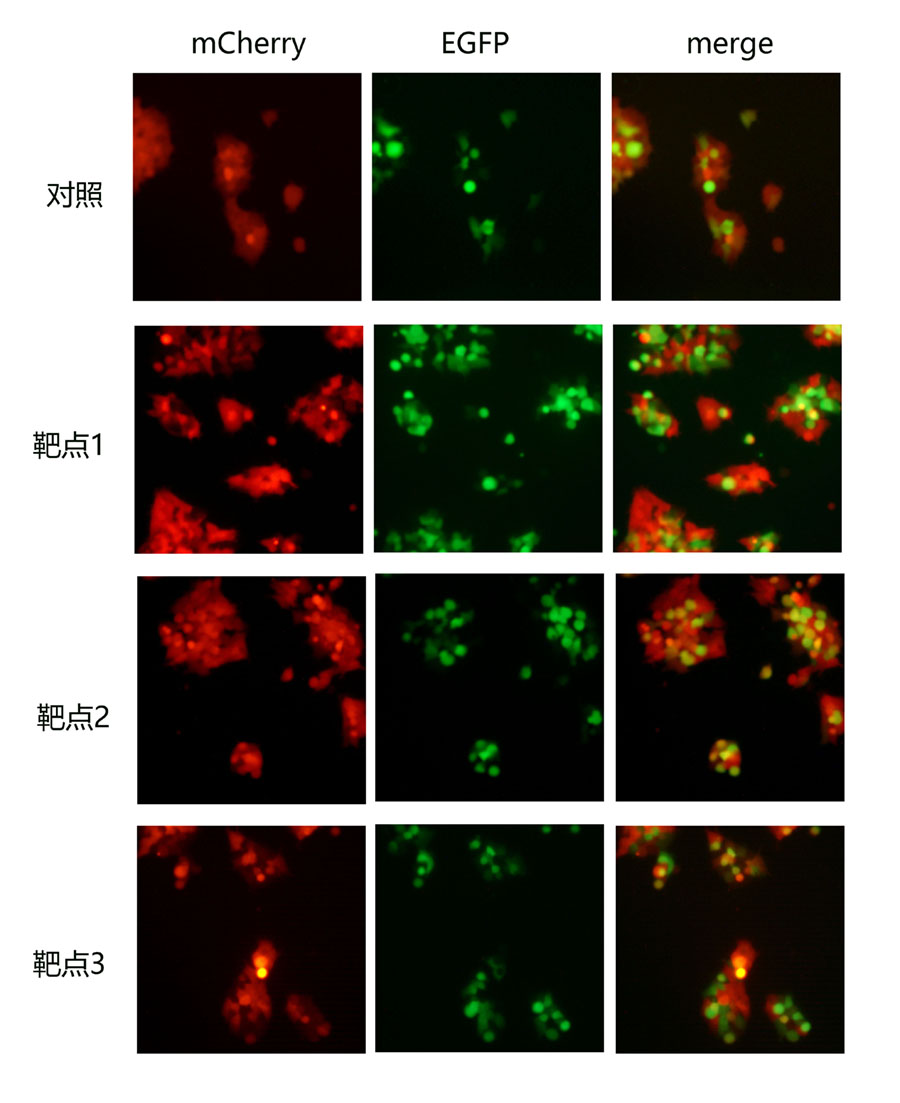
图5 荧光显微镜检测基因敲除效果。绿色荧光代表转染成功的细胞。红色荧光为敲除靶基因mCherry。对照组中绿色荧光分布与红色荧光分布重叠,表示单转Cas9nick-BE3没有基因敲除效果。靶点1和靶点3,绿色荧光和红色荧光镶嵌分布,显示较高的基因敲除效率。靶点2效果相对较差。
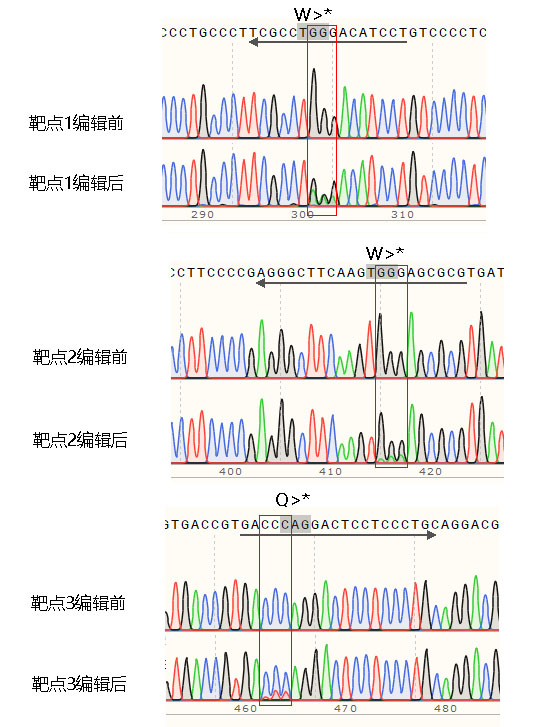
4.2、基因组PCR测序结果
5、结果分析
荧光检测结果和测序结果都显示转入的基因编辑质粒,在3个靶点位置都实现了mcherry基因敲除。从荧光结果和测序结果看,靶点1的敲除效率相对较高。我们观察到靶点2在体外酶切实验中切割效率较差,在细胞实验中,靶点2的编辑效果也是最差的。之前在需要双链DNA断裂的基因编辑实验中已经观测到,靶点的体外酶切实验结果与DSB基因编辑实验结果有较大相关性。本次实验中,靶点的体外酶切实验与细胞中mcherry基因敲除的结果也基本一致。我们将在后续的iStop基因敲除实验前加入体外酶切靶点验证,以进一步确认相关性是否广泛存在。
本次实验中,我们在转染后未进行任何细胞筛选,在转染后72小时直接进行检测。在靶点1组,所有几乎所有表达绿色荧光的细胞,我们都观察到mcherry荧光减弱或者消失,显示出极高的基因敲除效率。考虑到293V-cherry细胞是挑选的mcherry高表达单克隆细胞株,mcherry基因在基因组中极有可能为多个拷贝,这样的敲除效率就更加令人惊叹。iStop基因敲除是我们验证过的效率最高,使用最方便的基因敲除方法。
在设计mcherry靶点时,我们感觉要在指定的区域挑选出合适的靶点并不容易。特定的几个密码子必须位于距离PAM序列NGG特定的位置范围内。如果 序列较短,可能会遇到没有可用靶点的问题。好在Walton等人在后续的研究中(2),通过放松PAM限制,几乎实现了无需PAM实现Cas9蛋白对靶点的识别。我们也做了无PAM限制的Cas9突变体进行iStop基因敲除的试验,有的靶点效果也很理想。但是目前对无PAM限制的Cas9突变体特性了解不多,对靶点的预测更加困难,我们正在着力解决这一问题。
参考文献
1、Billon, Pierre et al. “CRISPR-Mediated Base Editing Enables Efficient Disruption of Eukaryotic Genes through Induction of STOP Codons.” Molecular cell vol. 67,6 (2017): 1068-1079.e4. doi:10.1016/j.molcel.2017.08.008
2、Walton, Russell T et al. “Unconstrained genome targeting with near-PAMless engineered CRISPR-Cas9 variants.” Science (New York, N.Y.) vol. 368,6488 (2020): 290-296. doi:10.1126/science.aba8853