023-67630383
 销售咨询QQ2548969917
销售咨询QQ2548969917 技术咨询QQ1291769782
技术咨询QQ1291769782热线/微信:13681365274
 销售咨询QQ2548969917
销售咨询QQ2548969917 技术咨询QQ1291769782
技术咨询QQ1291769782微信扫一扫
下载说明书


CRISPR-Cas系统为基础研究和临床转化应用中正展现出强大的实力和无穷的潜力,其中应用最为广泛的SpCas9系统能与sgRNA形成复合物切割靶位点引入DNA双链断裂(DSBs),之后由非同源重组修复(NHEJ)途径诱导插入缺失突变,或在DNA修复模板的存在下通过同源重组修复(HDR)途径实现精准基因编辑。
此外,以CRISPR-Cas9为基础的各种新工具也不断涌现,其中,通过融合不同脱氨基酶获得的单碱基编辑系统CBE(Cytosine Base Editor)和ABE(Adenine Base Editor),可分别实现C·G--T·A碱基对的转换和A·T--G·C碱基对的转换,这对上述的近半数点突变遗传病的治疗有重要意义,因而在基因治疗领域的应用前景相当广阔。不过,CRISPR-Cas系统对靶位点的识别受限于特定的PAM识别序列:以SpCas9为例,其仅能识别编辑PAM序列为NGG的基因组位点,因而极大的限制了其应用。
长期以来,研究者们致力于优化升级Cas9蛋白,以拓展其对不同PAM序列的兼容性,并期望有朝一日能摆脱PAM序列困扰,让Cas9拥有全基因组范围内的编辑能力。以SpCas9为例:Keith Joung实验室最早于2015年便通过易错PCR策略获得可识别NGA的SpCas9-VRQR突变体及NGCG的SpCas9-VRER突变体【1】。2018年,David Liu实验室利用其独有的定向演化技术PACE构建出可识别NGG、NG、GAA和GAT的xCas9 3.7变体【2】;同年Nureki实验室构建出活性更强的SpCas9-NG变体,其识别的PAM序列拓展至NG【3】。2020年,David Liu实验室利用PACE技术更进一步,其构建出的一系列SpCas9突变体识别的PAM序列拓展至NRNH(R为A/G, H为A/C/T),这一系列的工作让SpCas9及其突变体几乎摆脱了PAM困扰【4】(图1)。

2020年3月27日,来自哈佛医学院和麻省总医院的Benjamin P. Kleinstiver(【1】的一作)实验室在Science杂志上发表了题为Unconstrained genome targeting with near-PAMless engineered CRISPR-Cas9 variants的论文。文章再度对SpCas9蛋白进行了强势升级,改造后的SpCas9突变体SpRY几乎完全摆脱了PAM困扰,其识别的PAM序列涵盖NRN和NYN(Y为C/T)(NRN > NYN)。SpRY和以此为基础构建的单碱基编辑系统在PAM为NRN的位点处展现出强大的编辑能力,而在PAM为NYN的位点处的编辑能力虽然有所降低,但依然可观。

根据SpCas9的结构特征以及2015年报道的可识别PAM序列为NGA的SpCas9-VRQR突变体(D1135V/G1218R/R1335Q/T1337R),研究者推测包含R1335Q突变及其它位于PI结构域的突变是拓展PAM识别位点的基础,因此研究者将在SpCas9-VRQR的基础上进一步拓展PAM识别位点。
为研究PI结构域各类点突变对PAM识别位点的影响,研究者设计出一种高通量PAM分析法HT-PAMDA以系统性分析SpCas9突变体文库的PAM偏好性。通过HT-PAMDA分析,研究者发现一种突变体SpG(D1135L/S1136W/G1218K/E1219Q/R1335Q/T1337R) 可识别的PAM序列为NG,其对NGA、NGC、NGT和NGG并无明显偏好性。
而后,针对PAM序列为NGNN的78种位点,研究者通过细胞实验对野生型SpCas9、xCas9 3.7、SpCas9-NG和SpG的PAM偏好性和编辑活性进行比较,发现SpG在各类PAM序列处的表现最为稳定。此外,以SpG为基础构建的单碱基编辑系统SpG-CBE和SpG-ABE在各类PAM序列处的单碱基编辑活性也最为稳定。
从结构上而言,R1333Q突变或可使得SpCas9识别PAM序列为NAN的位点,然而SpG(R1333Q)在PAM序列为NRN的位点处的编辑活性甚微;不过研究者发现,L1111R和A1322R突变可恢复SpG(R1333Q)的部分编辑活性。
随后通过结构分析、HT-PAMDA实验和细胞实验,研究者发现SpG(L1111R/A1322R)+A61R/N1317R/R1333P在PAM序列为NRN和部分NYN位点处拥有最佳的编辑活性,该突变体也被命名为SpRY。而后,研究者通过细胞实验对SpRY及相关的单碱基编辑系统SpRY-CBE和SpRY-ABE在各类不同的PAM序列处的编辑活性进行系统分析,证实SpRY的编辑能力几乎摆脱了PAM困扰,其识别的PAM序列特征为NNN(NRN > NYN)(图2)。
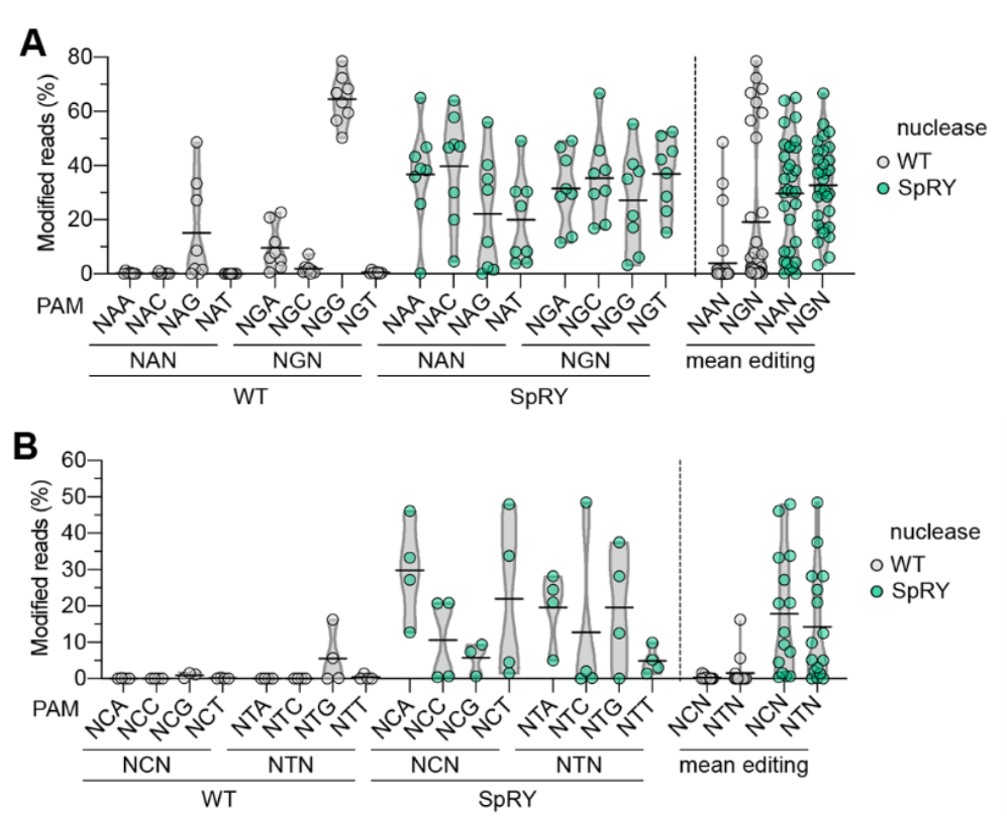
之后,研究者还证实,高保真版本的SpCas9-HF1突变体可以和SpG及SpRY兼容,从而获得不受PAM限制且安全性高的SpG-HF1和SpRY-HF1突变体。有了如此强大的SpG和SpRY工具及衍生的SpG/SpRY-CBE及SpG/SpRY-ABE,研究者能更加自如精准的诱导各种点突变。
总体而言,本研究开发的SpCas9突变体SpRY是当前对PAM序列兼容性最高的SpCas9突变体,几乎完全摆脱了PAM序列的限制,其在基因组范围内的编辑能力得到了极大的提高,而衍生而来的单碱基编辑系统让精准编辑几乎拓展至全基因组范围。
原文链接https://science.sciencemag.org/
相关产品
SpCas9突变体SpRY-NLS蛋白 spCas9 SpRY突变体体外酶切试剂盒
content/early/2020/03/25/science.aba8853